
 首页
首页
Genome tracing工具可根据新冠病毒基因组或突变数据计算出其在数据库中亲缘关系最近的序列,并显示其在全球时间空间的分布。

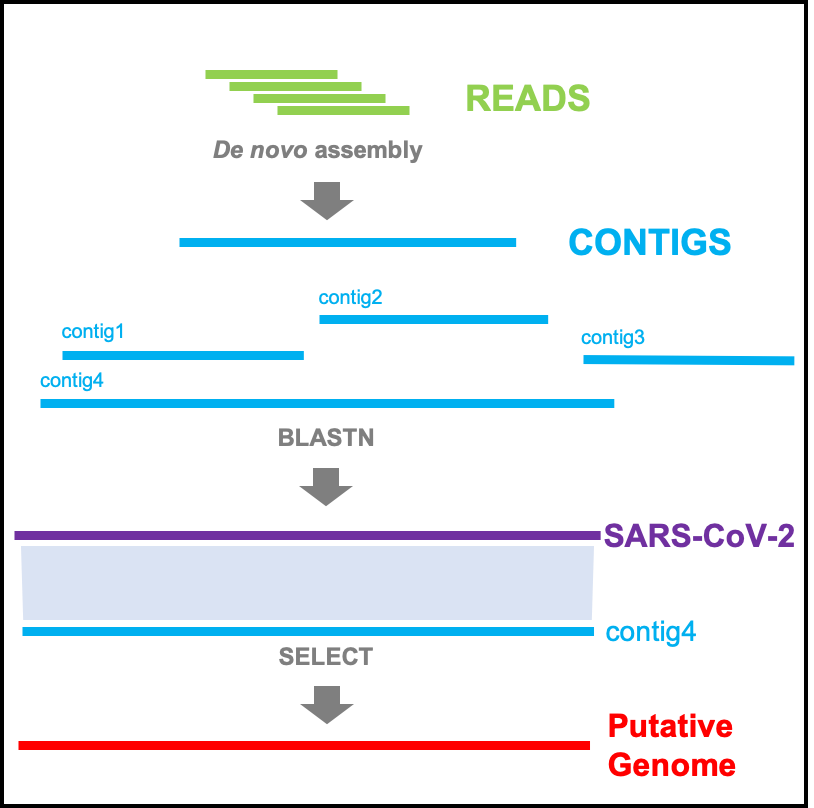
Denovo Assembly工具可对二代测序原始数据进行序列拼接,并检测样本组装结果中含有的新冠病毒序列。
BLAST工具可对用户在线提交的核酸序列进行BLAST比对,可选择新冠参照基因组、新冠序列数据库、冠状病毒基因组数据库。

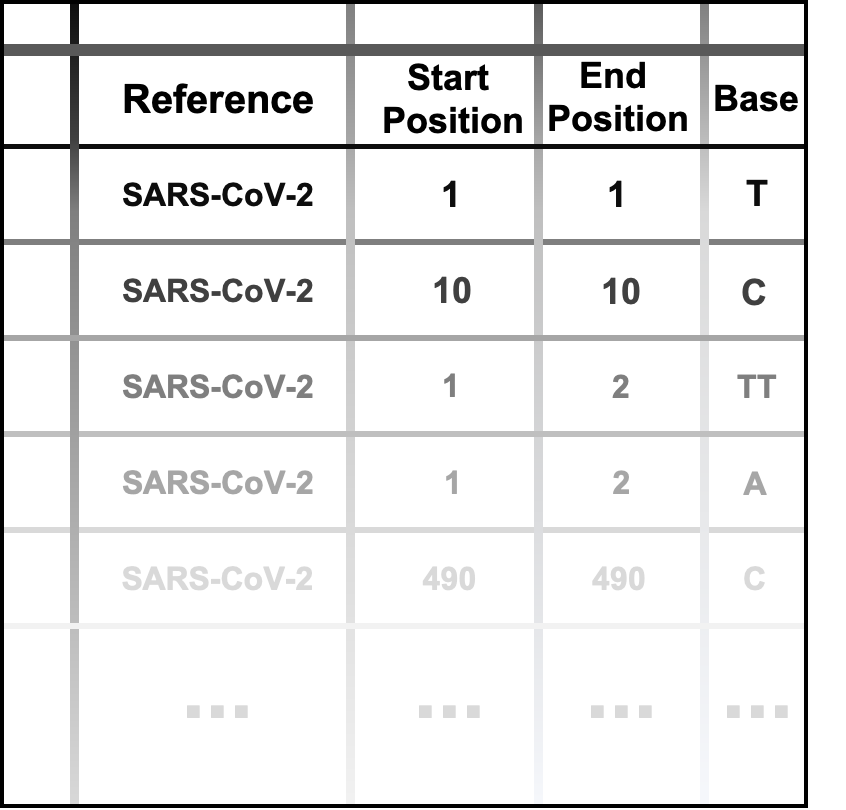
Genome-to-Variants工具可将用户提交的新冠病毒基因组序列与参照基因组进行比对,检测SNP/indel变异位点。

Variant Annotation工具用户输入的变异位点进行功能注释,显示变异位点所在基因、密码子和氨基酸的改变。
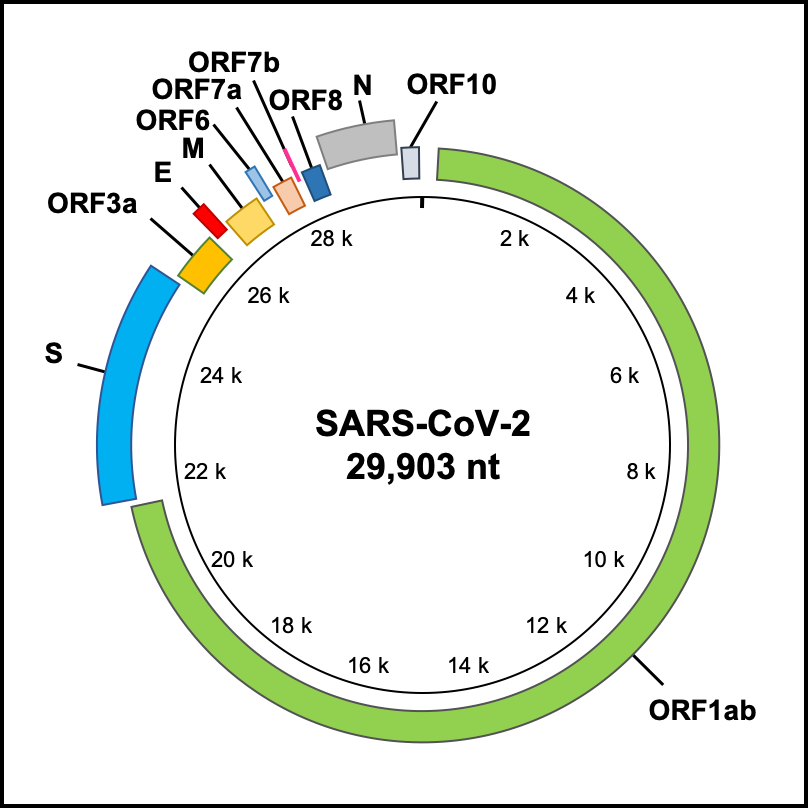
Genome Annotation工具可对用户提交的新冠病毒序列进行基因注释。
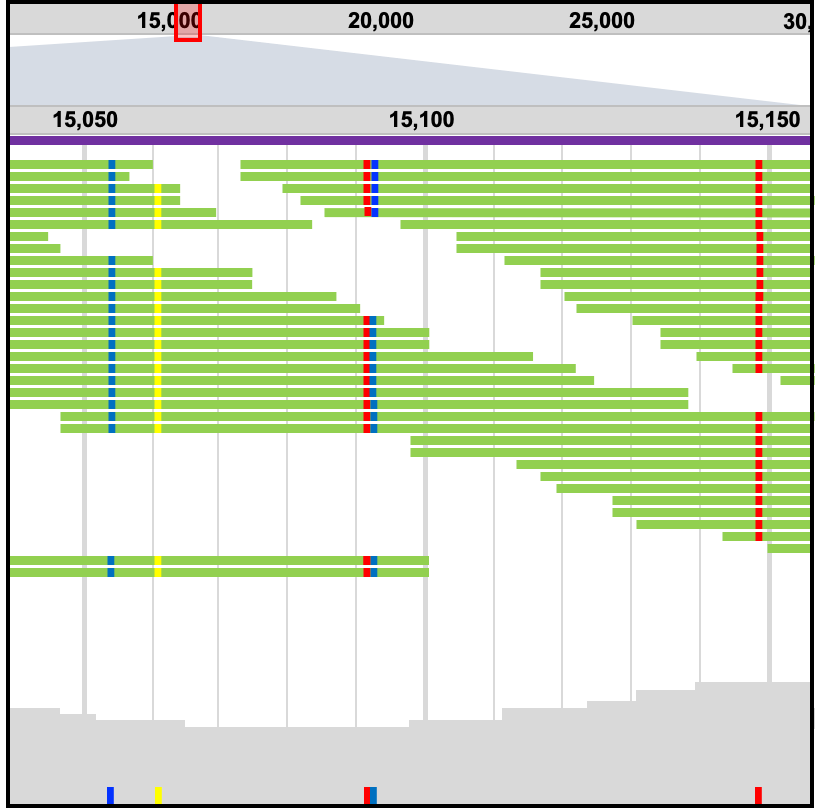
Fastq-to-Variants工具可将二代测序数据与新冠病毒基因组进行序列比对,检测SNP/indel变异位点并进行功能注释。
SeqQC可以对FASTQ格式和BAM格式的文件进行序列质量评估,它是使用FastQC和MultiQC程序进行计算的。

单倍型网络构建工具可以实现对用户上传的变异数据文件和meta数据文件结合我们库中已有数据进行整体网络图的计算和展示。
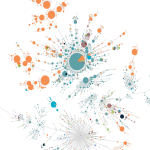
使用最大似然法构建系统发育树。该工具可将用户提交的序列使用IQ-TREE或RAxML构建系统发育树。
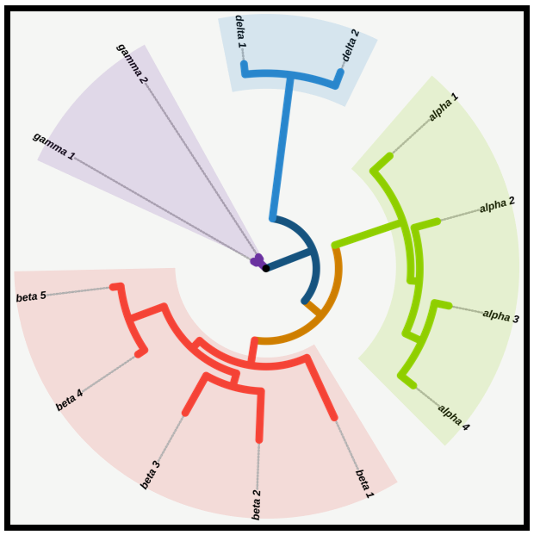
该工具可使用Pangolin软件进行谱系预测,并将确认为新冠病毒的序列使用RAxML-EPA算法放置于新冠病毒参考系统演化树进行可视化展示。
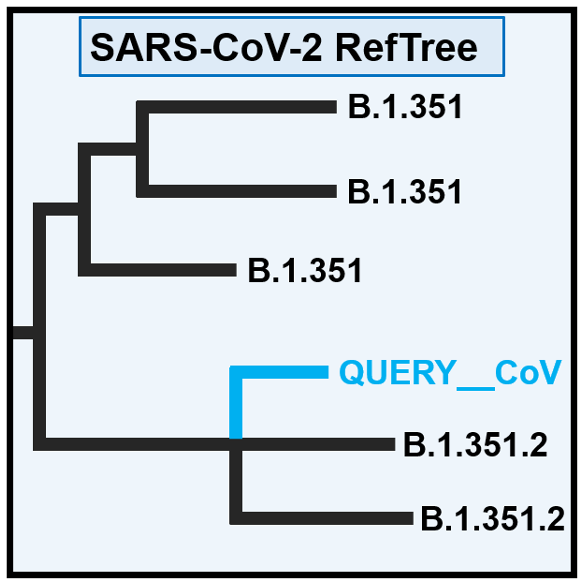
Pangolin COVID-19 Lineage Assigner 可将用户提交的序列使用Pangolin软件进行Pango谱系预测。
